乙肝病毒(HBV)感染是我國(guó)乃至全世界的一個(gè)嚴(yán)重的公共衛(wèi)生問(wèn)題���,是導(dǎo)致肝炎的主要原因之一���,長(zhǎng)期感染會(huì)導(dǎo)致肝纖維化、肝硬化甚至肝癌的發(fā)生,目前全球慢性HBV感染者約有2.5億人�。HBV基因組DNA是一個(gè)松弛環(huán)狀的DNA(rcDNA),長(zhǎng)度約3.2 kb��。一旦感染細(xì)胞��,rcDNA會(huì)進(jìn)入到細(xì)胞核并轉(zhuǎn)化為共價(jià)閉合環(huán)狀DNA(cccDNA)���,cccDNA是HBV所有病毒基因轉(zhuǎn)錄的模板�����,以微染色體的形式穩(wěn)定存在且難以清除�,是HBV反復(fù)復(fù)發(fā)且難以清除的根本原因[1, 2], 因此研究HBV cccDNA轉(zhuǎn)錄調(diào)控的分子機(jī)制具有重要的科研價(jià)值和臨床研究意義��。
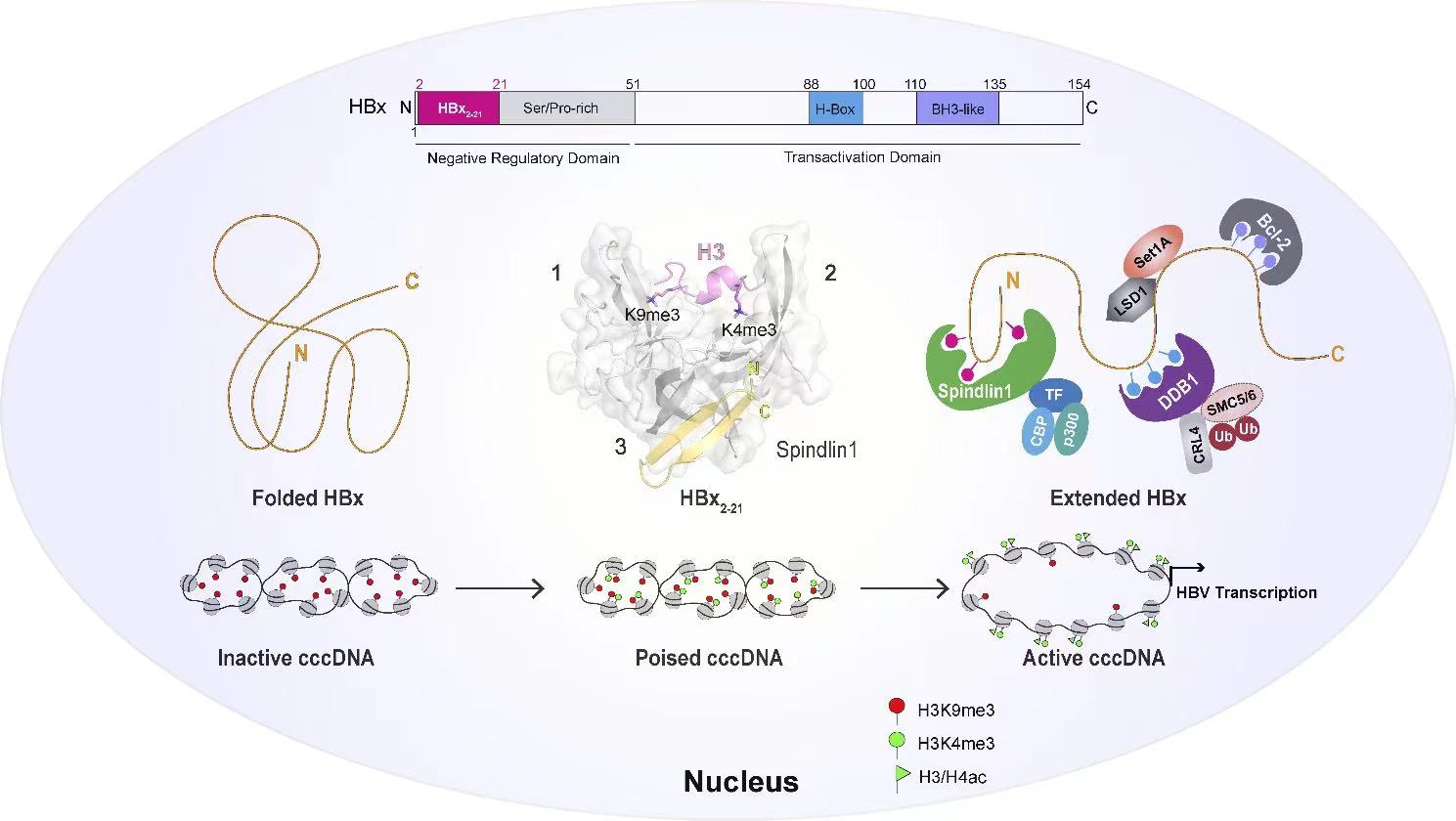
2023年8月3日,清華大學(xué)醫(yī)學(xué)院李海濤課題組��、北京生命科學(xué)研究所/清華大學(xué)生物醫(yī)學(xué)交叉研究院李文輝課題組和清華大學(xué)醫(yī)學(xué)院王大亮副教授合作�,在Nature Communications雜志在線發(fā)表了題為"Molecular insights into Spindlin1-HBx interplay and its impact on HBV transcription from cccDNA minichromosome"(Spindlin1-HBx互作及其影響乙肝病毒閉合環(huán)狀DNA微染色體轉(zhuǎn)錄的分子機(jī)理解析)的研究論文,報(bào)道了HBV編碼的關(guān)鍵調(diào)控蛋白HBx如何通過(guò)劫持和利用表觀因子Spindlin1蛋白來(lái)攻克異染色質(zhì)障礙進(jìn)而促進(jìn)HBV cccDNA轉(zhuǎn)錄的分子機(jī)理��。
該研究通過(guò)基于表面等離子共振成像(SPRi)技術(shù)的多肽陣列篩選[3]���、等溫量熱滴定(ITC)和結(jié)構(gòu)生物學(xué)等方法發(fā)現(xiàn)HBx的N端保守序列(第2-21位的氨基酸�����,HBx2-21)可以與Spindlin1蛋白的第3個(gè)Tudor結(jié)構(gòu)域互作���,并解析了Spindlin1-HBx2-21復(fù)合物的晶體結(jié)構(gòu)。接著�����,通過(guò)結(jié)構(gòu)分析����、生化結(jié)合以及細(xì)胞實(shí)驗(yàn)發(fā)現(xiàn),HBx蛋白與Spindlin1第3個(gè)Tudor的結(jié)合可以與Spindlin1第1和第2個(gè)Tudor結(jié)構(gòu)域?qū)M蛋白甲基化修飾的識(shí)別很好的共存�。有意思的是����,體外ITC實(shí)驗(yàn)表明與HBx結(jié)合后�,Spindlin1對(duì)組蛋白H3“K4me3-K9me3”二價(jià)修飾的識(shí)別能力提高了3.5倍�����,親和力高達(dá)3.8 納摩爾��,是迄今報(bào)導(dǎo)的最強(qiáng)組蛋白修飾識(shí)別事件��。接著����,研究者利用李文輝教授課題組發(fā)現(xiàn)并建立的HBV感染HepG2-NTCP細(xì)胞模型[4, 5]開(kāi)展了一系列功能實(shí)驗(yàn)。研究表明Spindlin1-HBx互作可以促進(jìn)HBV cccDNA的轉(zhuǎn)錄���,并且這種促進(jìn)作用與HBV cccDNA微染色體從H3K9me3標(biāo)志的異染色質(zhì)狀態(tài)向以H3K4me3和乙?��;揎棙?biāo)志的活躍染色質(zhì)狀態(tài)的轉(zhuǎn)變有關(guān)。值得一提的是�����,該研究還發(fā)現(xiàn)Spindlin1與HBx的N端保守序列互作會(huì)影響HBx蛋白構(gòu)象的改變,將HBx構(gòu)象從折疊的不活躍狀態(tài)轉(zhuǎn)變?yōu)樯煺沟幕钴S狀態(tài)����,進(jìn)而促進(jìn)HBx與其它互作因子如DDB1 [6]和Bcl-2 [7]等的結(jié)合來(lái)協(xié)同調(diào)控HBV基因組的活化。
該研究揭示了Spindlin1與HBx蛋白互作的結(jié)構(gòu)基礎(chǔ)�����,闡述了Spindlin1-HBx通過(guò)調(diào)控HBV cccDNA修飾狀態(tài)及HBx蛋白構(gòu)象改變進(jìn)而促進(jìn)HBV轉(zhuǎn)錄的分子機(jī)理��。李海濤課題組致力于揭示表觀調(diào)控因子在遺傳信息解讀�,以及癌癥、病毒和白血病等人類疾病發(fā)生中的作用機(jī)理����。在此前的報(bào)道中,李海濤課題組發(fā)現(xiàn)表觀調(diào)控因子Spindlin1可以通過(guò)其第1和第2個(gè)Tudor結(jié)構(gòu)域識(shí)別多種組蛋白甲基化修飾模式����,響應(yīng)不同的上游甲基化“書(shū)寫(xiě)器”(writer)通路,識(shí)別不同基因組區(qū)段的H3K4me3修飾或修飾組合景觀�����,從表觀遺傳層面調(diào)控重要基因的表達(dá)[8, 9]。本研究闡明了Spindlin1蛋白3個(gè)Tudor結(jié)構(gòu)域可以協(xié)同作用共同促進(jìn)HBV cccDNA的轉(zhuǎn)錄��,拓寬了領(lǐng)域內(nèi)對(duì)表觀調(diào)控因子在HBV生命周期中發(fā)揮的特異作用機(jī)制的理解���,為靶向HBV慢性感染及相關(guān)疾病的治療提供了重要理論基礎(chǔ)��。
清華大學(xué)醫(yī)學(xué)院李海濤教授����、北京生命科學(xué)研究所/清華大學(xué)生物醫(yī)學(xué)交叉研究院李文輝教授和清華大學(xué)醫(yī)學(xué)院王大亮副教授為共同通訊作者���,清華大學(xué)醫(yī)學(xué)院李海濤課題組博士后劉偉和北京生命科學(xué)研究所已畢業(yè)博士生姚奇艷為本文的共同第一作者,清華大學(xué)醫(yī)學(xué)院已出站博士后蘇曉楠和醫(yī)學(xué)實(shí)驗(yàn)班2017級(jí)學(xué)生鄧雅方等人參與了本研究��。前中科院納米所研究員朱勁松博士及其博士生楊墨在基于SPRi的多肽陣列篩選實(shí)驗(yàn)中提供了重要技術(shù)支撐����。
該工作得到國(guó)家自然科學(xué)基金委員會(huì)、國(guó)家重點(diǎn)研發(fā)計(jì)劃和北京市重大科技專項(xiàng)的資助�。李海濤教授是清華-北大生命科學(xué)聯(lián)合中心、北京生物結(jié)構(gòu)前沿研究中心�、分子腫瘤學(xué)全國(guó)重點(diǎn)實(shí)驗(yàn)室、教育部蛋白質(zhì)科學(xué)重點(diǎn)實(shí)驗(yàn)室���,以及山西醫(yī)科大學(xué)-清華大學(xué)醫(yī)學(xué)院前沿醫(yī)學(xué)協(xié)同創(chuàng)新中心成員��。
原文鏈接:
https://www.nature.com/articles/s41467-023-40225-w
參考文獻(xiàn):
參考文獻(xiàn)
1. Bock, C.T., et al., Hepatitis-B Virus Genome Is Organized into Nucleosomes in the Nucleus of the Infected Cell. Virus Genes, 1994. 8(3): p. 215-229.
2. Nassal, M., HBV cccDNA: viral persistence reservoir and key obstacle for a cure of chronic hepatitis B. Gut, 2015. 64(12): p. 1972-84.
3. Zhao, S., et al., Kinetic and high-throughput profiling of epigenetic interactions by 3D-carbene chip-based surface plasmon resonance imaging technology. Proc Natl Acad Sci U S A, 2017. 114(35): p. E7245-e7254.
4. Sun, Y., et al., NTCP-Reconstituted In Vitro HBV Infection System. Methods Mol Biol, 2017. 1540: p. 1-14.
5. Yan, H., et al., Sodium taurocholate cotransporting polypeptide is a functional receptor for human hepatitis B and D virus. Elife, 2012. 1: p. e00049.
6. Decorsiere, A., et al., Hepatitis B virus X protein identifies the Smc5/6 complex as a host restriction factor. Nature, 2016. 531(7594): p. 386-9.
7. Jiang, T., et al., Structural and biochemical analysis of Bcl-2 interaction with the hepatitis B virus protein HBx. Proc Natl Acad Sci U S A, 2016. 113(8): p. 2074-9.
8. Su, X., et al., Molecular basis underlying histone H3 lysine-arginine methylation pattern readout by Spin/Ssty repeats of Spindlin1. Genes Dev, 2014. 28(6): p. 622-36.
9. Zhao, F., et al., Molecular basis for histone H3 "K4me3-K9me3/2" methylation pattern readout by Spindlin1. J Biol Chem, 2020. 295(49): p. 16877-16887.